La levure Saccharomyces cerevisiae est l’un des organismes les plus importants dans la biotechnologie. Outre son rôle dans le brassage et la fabrication du pain, la levure a été utilisé comme un organisme hôte pour la production de produits pharmaceutiques importants à partir de gènes clonés. Dans un premier temps le développement des vecteurs de clonage de la levure, a été stimulée, par la découverte d’un plasmide qui est présent dans la plupart des souches de S. cerevisiae. Le 2 µ plasmide, comme on l’appelle, est l’un des rares plasmides trouvés dans les cellules eucaryotes.
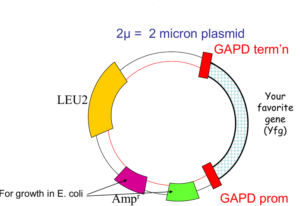
Les marqueurs sélectionnables pour le plasmide 2 µm:
Le plasmide de 2 µm est un excellent élément pour un vecteur de clonage. Il a une taille de 6 kb, ce qui est idéal pour un vecteur et existe dans la cellule de levure à un nombre de copies compris entre 70 et 200. La réplication utilise une origine de plasmide, plusieurs enzymes fournies par la cellule hôte et les protéines codées Par les gènes REP1 et REP2 portés par le plasmide. Cependant, tout n’est pas parfaitement simple dans l’utilisation du plasmide de 2 μm comme vecteur de clonage. Tout d’abord, il y a la question d’un marqueur sélectionnable. Certains vecteurs de clonage de levure portent des gènes conférant une résistance aux inhibiteurs tels que le méthotrexate et le cuivre, mais la plupart des vecteurs de levure populaires utilisent un type de sélection radicalement différent.
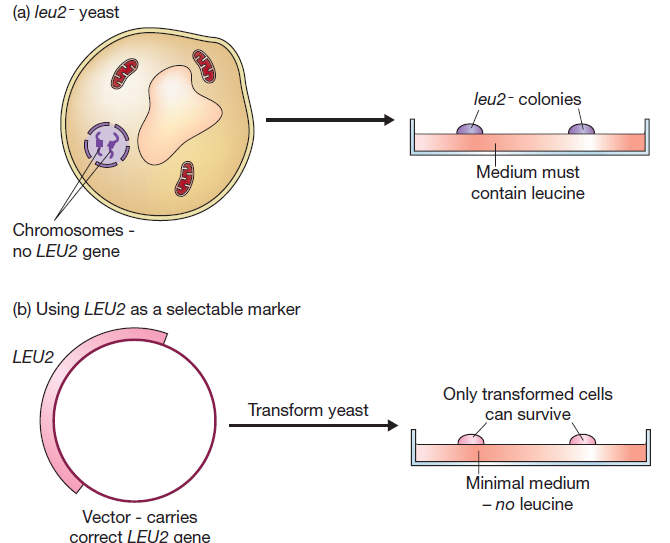
En pratique, on utilise un gène de levure normal, généralement celui qui code pour une enzyme impliquée dans la biosynthèse des acides aminés. Un exemple est le gène LEU2, qui code pour la b-isopropyl-malate déshydrogénase, l’une des enzymes impliquées dans la conversion de l’acide pyruvique en leucine. Afin d’utiliser le LEU2 comme marqueur de sélection, un type particulier d’organisme hôte est nécessaire. L’hôte doit être un mutant auxotrophique qui possède un gène LEU2 non fonctionnel. Une telle levure leu2- est incapable de synthétiser la leucine et ne peut survivre que si cet acide aminé est fourni comme nutriment dans le milieu de croissance . La sélection est possible parce que les transformants contiennent une copie du gène LEU2 supportée par un plasmide et sont ainsi capables de se développer en l’absence de l’acide aminé. Dans une expérience de clonage, les cellules sont étalées sur un milieu minimal, qui ne contient pas d’acides aminés ajoutés. Seules les cellules transformées sont capables de survivre et de former des colonies.
Les vecteurs basés sur le plasmide 2 µm – les plasmides épisomiques :
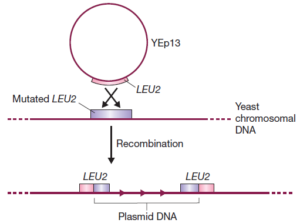
Les vecteurs dérivés du plasmide de 2 μm sont appelés plasmides épisomiques de levure (YEps=yeast episomal plasmids). Quelques YEps contiennent le plasmide de 2 µm en entier, d’autres incluent juste l’origine de réplication de 2 μm. Un exemple de ce dernier type est YEp13. YEp13 illustre plusieurs caractéristiques générales de vecteurs de clonage de levure. Tout d’abord, il s’agit d’un vecteur navette. Outre l’origine de replication de 2 μm et le gène LEU2 sélectionnable, YEp13 inclut également la séquence entière de pBR322, et peut donc se répliquer et être sélectionné pour à la fois dans la levure et dans E. coli.
Il ya plusieurs lignes de raisonnement derrière l’utilisation des vecteurs navette. L’une est qu’il pourrait être difficile de récupérer la molécule d’ADN recombinant d’une colonie de levure transformée. Ce n’est pas un tel problème avec YEps, qui sont présents dans les cellules de levure principalement sous forme de plasmides, mais avec d’autres vecteurs de levure, qui peuvent s’intégrer dans un des chromosomes de levure, la purification pourrait être impossible. Ceci est un inconvénient car, dans de nombreuses expériences de clonage, la purification de l’ADN recombinant est essentielle pour que la construction correcte soit identifiée par exemple par séquençage d’ADN. La procédure standard lors du clonage dans la levure est donc d’effectuer l’expérience de clonage initiale avec E. coli et de sélectionner des recombinants dans cet organisme. Les plasmides recombinants peuvent ensuite être purifiés, caractérisés et la molécule correcte Introduite dans la levure.
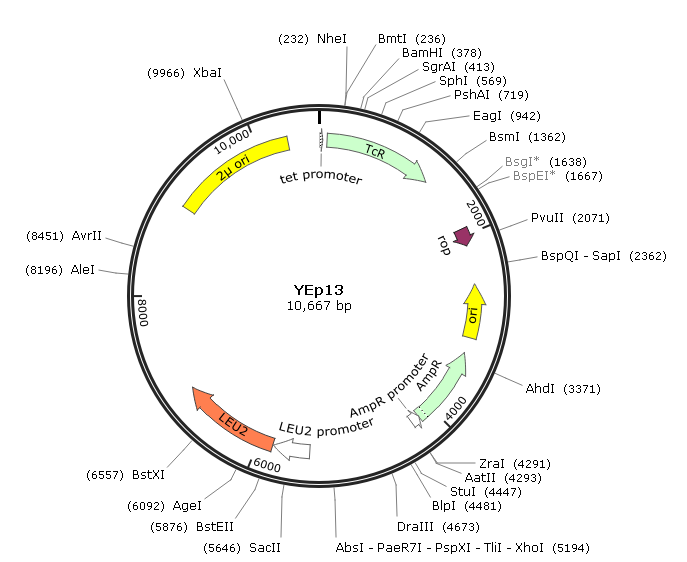
Un YEp peut s’insérer dans l’ADN chromosomique de levure
Le mot «épisomale» indique qu’un YEp peut se répliquer en tant que plasmide indépendant, mais implique également l’intégration dans l’un des chromosomes de levure (voir la définition de «épisome»). L’intégration se produit parce que le gène porté sur le vecteur comme marqueur de sélection est très similaire à la version mutante du gène présent dans l’ADN chromosomique de levure. Avec YEp13, par exemple, une recombinaison homologue peut se produire entre le gène du plasmide LEU2 et le gène LEU2 du mutant de levure, provoquant l’insertion du plasmide entier dans l’un des chromosomes de levure. le Plasmide peut rester intégré, ou un événement de recombinaison ultérieur peut l’excisé de nouveau.
 Autres types de vecteur de clonage de levure :
Autres types de vecteur de clonage de levure :
En plus des YEps , il existe plusieurs autres types de vecteurs de clonage à utiliser avec S.cerevisiae. Les deux plus importants sont les suivantes:
A) Les plasmides intégrés dans la levure (YIps = Yeast integrative plasmids ) sont essentiellement des plasmides bactériens portant un gène de levure. Un exemple est YIp5, qui est le pBR322 avec un gène URA3 inséré.
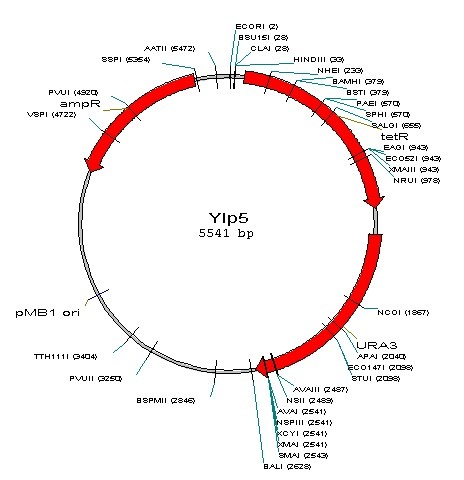
Ce gène code pour l’orotidine-5′-phosphate décarboxylase (une enzyme qui catalyse une des étapes de la voie de biosynthèse pour les nucleotides pyrimidiques) et est utilisée comme marqueur sélectionnable exactement de la même manière que LEU2. Un YIp ne peut pas se répliquer en tant que plasmide car il ne contient aucune partie du plasmide 2 µm et dépend plutôt pour sa survie de son intégration dans l’ADN chromosomique de levure. L’intégration se produit juste comme décrit pour un YEp.
B) Plasmides réplicatifs de levure (YRps=Yeast replicative plasmids )
Ils sont capables de se multiplier en tant que plasmides indépendants parce qu’ils portent une séquence d’ADN chromosomique qui comprend une origine de réplication. Les origines de réplication sont connues pour être situées très près de plusieurs des gènes de levure, comprenant un ou deux qui peuvent être utilisés comme marqueurs sélectionnables. YRp7 est un exemple de plasmide réplicatif. Il est composé de pBR322 plus le gène de levure TRP1. Ce gène, impliqué dans la biosynthèse du tryptophane, est situé adjacent à une origine chromosomique de réplication.
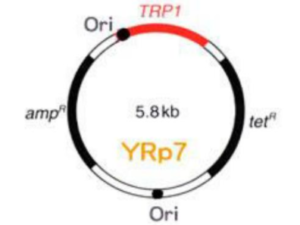
Le fragment d’ADN de levure présent dans YRp7 contient TRP1 et l’origine de réplication. Trois facteurs entrent en jeu pour décider quel type de vecteur de levure est le plus approprié pour une expérience de clonage particulière:
La première est la fréquence de transformation : une mesure du nombre de transformants qui peuvent être obtenus par microgramme de plasmidique. Une fréquence de transformation élevée est nécessaire si un grand nombre de recombinants sont nécessaires, ou si l’ADN de départ est insuffisant. YEps ont le plus haut fréquence de transformation, fournissant entre 10 000 et 100 000 cellules transformées Par µg. YRps sont également assez productifs, donnant entre 1000 et 10 000 transformants par µg, mais un YIp donne moins de 1000 transformants par µg et seulement 1-10 à moins que des procédures spéciales sont utilisées. La faible fréquence de transformation d’un YIp reflète que l’événement d’intégration chromosomique (qui est assez rare) est nécessaire avant que le vecteur puisse être conservé dans une cellule de levure.
Le deuxième facteur important est le nombre de copies. YEps et YRps ont le nombre de copie le plus élevé, 20-50 et 5-100, respectivement. En revanche, un YIp est habituellement présent à une seule copie par cellule. Ces chiffres sont importants si l’objectif est d’obtenir des protéines par le gène cloné, plus il y a de copies du gène, plus élevé sera le rendement attendu du produit protéique.
Alors pourquoi voudrait-on utiliser un YIp? La réponse est que YIps produit des recombinants très stables, comme la perte d’un YIp qui est devenu intégré dans un chromosome ne se produit qu’à une très faible fréquence. En revanche, les recombinants YRp sont extrêmement instables,les plasmides tendant à se rassembler dans la cellule mère lorsqu’une cellule fille se déclenche, la cellule fille n’est pas recombinante. Les recombinants YEp souffrent de problèmes similaires, bien qu’une meilleure compréhension de la biologie du plasmide de 2 µm ait permis de développer des YEps plus stable ces dernières années. Néanmoins, un YIp est le vecteur de choix si les besoins de l’expérience exigent que les cellules de levure recombinantes retiennent le gène cloné pendant de longues périodes en culture.
Les chromosomes artificiels peuvent être utilisés pour cloner de longs morceaux d’ADN dans des levures :
Le dernier type de vecteur de clonage de levure à considérer est le chromosome artificiel de levure (YAC), qui présente une approche totalement différente au clonage de gène. Le développement des YAC a été un spin-off de la recherche fondamentale sur la structure des chromosomes eucaryotes, le travail qui a identifié les composants clés d’un chromosome comme étant:
– le centromère, qui est nécessaire pour que le chromosome soit réparti correctement aux cellules filles pendant la division cellulaire;
– deux télomères, les structures aux extrémités d’un chromosome qui sont nécessaires pour que les extrémités soient correctement reproduites et qui empêchent également le chromosome d’être grignoté par les exonucléases;
– les origines de la réplication, qui sont les positions le long du chromosome à l’origine de la réplication de l’ADN, similaire à l’origine de la réplication d’un plasmide.
Une fois que la structure chromosomique a été définie de cette manière, la possibilité est apparue que les composants individuels puissent être isolés par des techniques d’ADN recombinant, puis rejoints ensemble dans le tube à essai, créant un chromosome artificiel. Comme les molécules d’ADN présentes dans les chromosomes naturels de levure ont plusieurs centaines de kilobases de longueur, il serait possible avec un chromosome artificiel de cloner de longs morceaux d’ADN.
La structure et l’utilisation d’un vecteur YAC
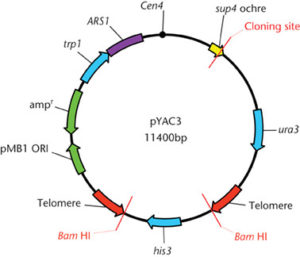
Plusieurs vecteurs YAC ont été développés mais chacun est construit selon le même schémas, le pYAC3 étant un exemple typique. À première vue, pYAC3 ne ressemble pas beaucoup à un chromosome artificiel mais en examinant de plus près ses caractéristiques uniques apparaissent. pYAC3 est essentiellement un plasmide pBR322 dans lequel un certain nombre de gènes de levure ont été insérés. Deux de ces gènes, URA3 et TRP1, ont déjà été rencontrés comme marqueurs sélectionnables pour YIp5 et YRp7, respectivement. Comme dans YRp7, le fragment d’ADN qui porte TRP1 contient également une origine de replication, mais dans pYAC3 ce fragment est étendu encore plus pour inclure la séquence appelée CEN4, qui est l’ADN provenant de la région centromère du chromosome 4. Le fragment (TRP1-origine- CEN4) contient donc deux des trois composants du chromosome artificiel. Le troisième composant, les télomères, est fourni par les deux séquences appelées TEL. Ce ne sont pas des séquences de télomères complètes, mais une fois à l’intérieur du noyau de levure, ils agissent comme des séquences d’ensemencement sur lesquelles les télomères seront construits. Il reste juste une autre partie de pYAC3 qui n’a pas été mentionnée: SUP4, qui est le marqueur sélectionnable dans lequel le nouvel ADN est inséré pendant l’expérience de clonage.
La stratégie de clonage avec pYAC3 est la suivante. Le vecteur est d’abord restreint avec une combinaison de BamHI et SnaBI, coupant la molécule en trois fragments. Le fragment flanqué de sites BamHI est jeté, laissant deux bras, chacun limité par une séquence TEL et un site SnaBI. L’ADN à cloner, qui doit avoir des extrémités émoussées (SnaBI est un cutter d’extrémité émoussée, reconnaissant la séquence TACGTA), est ligaturé entre les deux bras, produisant le chromosome artificiel.
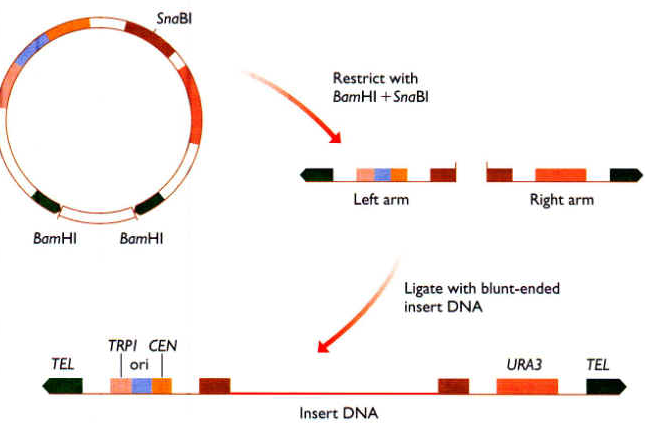
La transformation du protoplaste est alors utilisée pour introduire le chromosome artificiel dans S. cerevisiae. La souche de levure qui est utilisée est un double mutant auxotrope, trp1-ura3-, qui est converti en trp1 + ura3 + par les deux marqueurs sur le chromosome artificiel. Les transformants sont donc sélectionnés par culture sur milieu minimal, sur lequel seules des cellules contenant un chromosome artificiel correctement construit sont capables de se développer. Toute cellule transformée avec un chromosome artificiel incorrect, contenant deux bras droits ou deux bras gauches plutôt qu’un de chacun, n’est pas capable de croître sur un milieu minimal car l’un des marqueurs est absent. La présence de l’ADN d’insertion dans le vecteur peut être vérifiée en testant l’inactivation par insertion de SUP4, qui est réalisée par un simple test de couleur: les colonies blanches sont des recombinants, les colonies rouges ne le sont pas.
Utilisations des vecteurs YAC
Le stimulus initial dans la conception de chromosomes artificiels est venu de généticiens de levure qui voulait les utiliser pour étudier divers aspects de la structure et le comportement des chromosomes. Par exemple pour examiner la ségrégation des chromosomes lors de la méiose. Ces expériences ont établi que les chromosomes artificiels sont stables pendant la propagation dans des cellules de levure et ont soulevé la possibilité qu’ils pourraient être utilisés comme vecteurs pour des gènes qui sont trop longs pour être clones comme un seul fragment dans un vecteur d’E. coli. Plusieurs gènes de mammifères importants ont une longueur supérieure à 100 kb (par exemple, le gène de la mucoviscidose humaine est de 250 kb) qui est au-delà de la capacité de tous les systèmes de clonage d’E. Coli alors qu’ils sont dans la gamme d’un vecteur YAC. Les chromosomes artificiels de levure ont donc ouvert la voie à des études sur les fonctions et les modes d’expression de gènes qui avaient auparavant été intraitable à l’analyse par des techniques d’ADN recombinant. Une nouvelle dimension de ces expériences a été fournie par la découverte que, dans certaines circonstances, les YAC peuvent être propagés dans des cellules de mammifères, ce qui permet d’effectuer l’analyse fonctionnelle dans l’organisme dans lequel le gène réside normalement. Les chromosomes artificiels de levure sont également importants dans la production de banques de gènes. Rappelons qu’avec des fragments de 300 kb, la taille d’insert maximale pour le vecteur E. coli de capacité maximale, environ 30 000 clones sont nécessaires pour une banque de gènes humains. Cependant, les vecteurs YAC sont utilisés couramment pour cloner des fragments de 600 kb, et des types spéciaux sont capables de traiter l’ADN jusqu’à 1400 kb de longueur, ce dernier portant la taille d’une banque de gènes humains à seulement 6500 clones. Malheureusement, ces «méga-YAC» se heurtent à des problèmes de stabilité de l’insert, l’ADN cloné étant parfois réarrangé par recombinaison intramoléculaire. Néanmoins, les YAC ont été d’une immense valeur pour fournir de longues pièces d’ADN cloné pour une utilisation dans des projets de séquençage d’ADN à grande échelle.
Vecteurs pour d’autres levures et champignons :
Des vecteurs de clonage pour d’autres espèces de levures et de champignons sont nécessaires pour des études de base de la biologie moléculaire de ces organismes et pour étendre les utilisations possibles de levures et de champignons en biotechnologie. Les plasmides épisomiques basés sur le plasmide de 2 µm de S. cerevisiae sont capables de se répliquer dans quelques autres types de levures, mais la gamme des espèces n’est pas assez large pour que les 2 vecteurs µm soient de valeur générale. Dans tous les cas, les exigences de la biotechnologie sont mieux servies par des plasmides intégrateurs, équivalents à YIps, car ils fournissent des recombinants stables qui peuvent être cultivés pendant de longues périodes dans des bioréacteurs. Des vecteurs intégrateurs efficaces sont maintenant disponibles pour un certain nombre d’espèces, y compris des levures telles que Pichia pastoris et Kluveromyces lactis, et les champignons filamenteux tels que Aspergillus nidulans et Neurospora crassa.