Les enzymes agissant sur l’ADN peuvent être regroupées en quatre grandes classes, en fonction du type de réaction qu’elles catalysent:
1) Les nucléases sont des enzymes qui coupent, raccourcissent ou dégradent les molécules d’acide nucléique.
2) Les ligases rejoignent les molécules d’acide nucléique.
3) Les polymérases produisent des copies des molécules.
4) Les enzymes modificatrices éliminent ou ajoutent des groupes chimiques sur l’ADN.
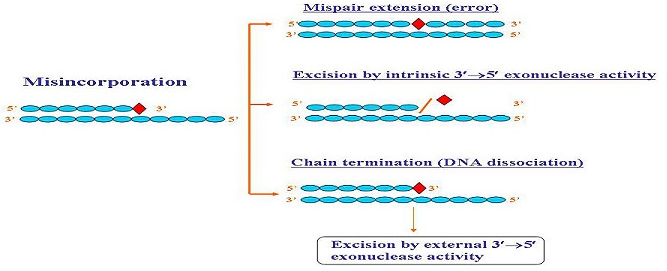
Avant d’examiner en détail chacune de ces classes d’enzymes, deux points doivent être signalés. La première est que, bien que la plupart des enzymes peuvent être assignés à une classe particulière, quelques-uns affichent des activités multiples qui s’étendent sur deux ou plusieurs classes. Plus important encore, de nombreuses polymérases combinent leur capacité à fabriquer de nouvelles molécules d’ADN avec une activité de dégradation de l’ADN associée (exemple, la nucléase).
Deuxièmement, il convient de noter qu’il existe de nombreuses enzymes similaires qui sont capables d’agir sur l’ARN. La ribonucléase utilisée pour éliminer l’ARN contaminant des préparations d’ADN en est un exemple. Bien que certaines enzymes agissant sur l’ARN aient des applications dans le clonage de gènes et seront mentionnées dans des chapitres ultérieurs, nous limiterons en général nos applications aux enzymes qui agissent sur l’ADN.
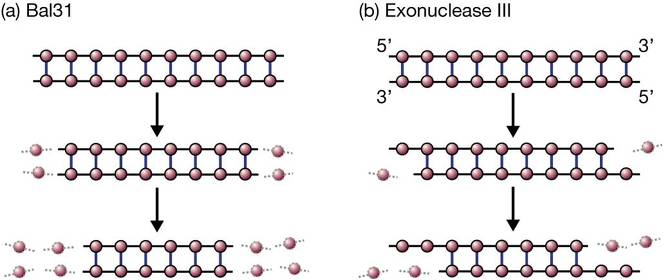
La principale distinction entre les différentes exonucléases réside dans le nombre de brins qui sont dégradés lorsqu’une molécule double brin est attaquée. L’enzyme appelée Bal31 (purifiée à partir de la bactérie Alteromonas espejiana) est un exemple d’exonucléase qui élimine les nucléotides des deux brins d’une molécule double brin. Plus la période de temps pendant laquelle Bal31 est autorisé à agir sur un groupe de molécules d’ADN, plus les fragments d’ADN résultant seront courts.
Le même critère peut être utilisé pour classer les endonucléases. L’endonucléase S1 (du champignon Aspergillus oryzae) ne clive que des brins simples, tandis que la désoxyribonucléase I (DNase I), préparée à partir de pancréas de vache, coupe les molécules simples et bicaténaires.
Les Nucléases :
Les nucléases dégradent les molécules d’ADN en brisant les liaisons phosphodiester qui lient un nucléotide à l’autre dans un brin d’ADN. Il existe deux types différents de nucléases :
1) Les exonucléases éliminent les nucléotides l’un après l’autre de l’extrémité d’une molécule d’ADN.
2) Les endonucléases sont capables de rompre les liaisons phosphodiester internes au sein d’une molécule d’ADN.
La principale distinction entre les différentes exonucléases réside dans le nombre de brins qui se dégradent lorsqu’une molécule double brin est attaquée. L’enzyme appelée Bal31 (purifiée à partir de la bactérie Alteromonas espejiana) est un exemple d’exonucléase qui élimine les nucléotides des deux brins d’une molécule double brin. Plus la période de temps pendant laquelle Bal31 est autorisé à agir sur un groupe de molécules d’ADN, plus les fragments d’ADN résultant seront courts.

En revanche, des enzymes telles que l’exonucléase III de E. coli dégradent seulement un brin d’une molécule double brin, laissant l’ADN monocaténaire comme produit.

Le même critère peut être utilisé pour classer les endonucléases. L’endonucléase S1 (du champignon Aspergillus oryzae) ne clive que des brins simples.

Et la désoxyribonucléase I (DNase I), préparée à partir de pancréas de vache, coupe les molécules simples et bicaténaires . La DNase I est non spécifique en ce qu’elle attaque l’ADN à n’importe quelle liaison phosphodiester interne, de sorte que le résultat final de l’action prologée de la DNase I est un mélange de mononucléotides et d’oligonucléotides très courts. Tandis que le groupe spécial d’enzymes appelées endonucléases de restriction ne clivent l’ADN double caténaire que sur un nombre limité de sites de reconnaissance spécifiques . Ces enzymes importantes sont décrites en détail dans un autre chapitre.

Les ligases :
Dans la cellule, la fonction de l’ADN ligase est de réparer les ruptures monocaténaires (« discontinuités ») qui surviennent dans des molécules d’ADN double brin pendant, par exemple, la réplication de l’ADN. Les ADN ligases de la plupart des organismes peuvent également recoller deux fragments individuels d’ADN double brin.
Les polymerases :
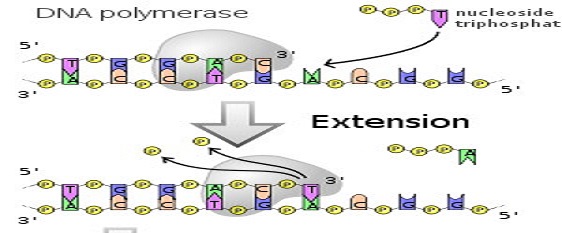
Les ADN polymérases sont des enzymes qui synthétisent un nouveau brin d’ADN complémentaire à un modèle d’ADN ou d’ARN existant. La plupart des polymérases ne peuvent fonctionner que si le gabarit possède une région à double brin qui sert d’amorce pour l’initiation de la polymérisation. Quatre types d’ADN polymérase sont utilisés couramment dans le génie génétique.
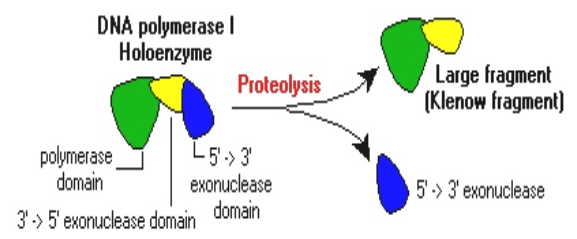
La première est l’ADN polymérase I, qui est habituellement préparée à partir d’ E. Coli. Cette enzyme s’attache à une courte région monocaténaire (ou entaille ) dans une molécule d’ADN principalement à double brin, puis synthétise un brin complètement nouveau, dégradant le brin existant à mesure qu’il progresse. L’ADN polymérase I est donc un exemple d’une enzyme avec une double activité, la polymérisation de l’ADN et la dégradation de l’ADN. Les activités de polymérase et de nucléase de l’ADN polymérase I sont contrôlées par différentes parties de la molécule d’enzyme.

L’activité nucléase est contenue dans les premiers 323 acides aminés du polypeptide, si bien que l’élimination de ce segment laisse une enzyme modifiée qui conserve la fonction polymérase mais est incapable de dégrader l’ADN. Cette enzyme modifiée, appelée fragment de Klenow, peut encore synthétiser un brin d’ADN complémentaire sur un modèle monocaténaire, mais comme il n’a pas d’activité nucléase, il ne peut pas poursuivre la synthèse une fois l’entaille rempli. Plusieurs autres enzymes, polymérases naturelles et des versions modifiées ont des propriétés similaires au fragment de Klenow. L’application principale de ces polymérases est dans le séquençage de l’ADN.

L’ADN polymérase Taq utilisée dans la réaction en chaîne par polymérase (PCR) est l’enzyme ADN polymérase I de la bactérie Thermus aquaticus. Cet organisme vit dans les sources chaudes, et beaucoup de ses enzymes, y compris l’ADN polymérase Taq, sont thermostables, ce qui signifie qu’ils sont résistants à la dénaturation par traitement thermique. C’est la particularité de Taq ADN polymérase qui la rend appropriée pour la PCR, car s’il n’était pas thermostable il serait inactivé lorsque la température de la réaction est élevée à 94 °C pour dénaturer l’ADN.

Le dernier type d’ADN polymérase qui est important dans le génie génétique est la transcriptase inverse, une enzyme impliquée dans la réplication de plusieurs types de virus. La transcriptase inverse est unique en ce qu’elle utilise comme modèle non l’ADN mais l’ARN. La capacité de cette enzyme à synthétiser un brin d’ADN complémentaire à un gabarit d’ARN est centrale à la technique appelée clonage d’ADN complémentaire (ADNc).

Les enzymes modifiant l’ADN :
Il existe de nombreuses enzymes qui modifient les molécules d’ADN par addition ou élimination de groupes chimiques spécifiques. Les plus importantes sont les suivantes:
(A) phosphatase alcaline: (à partir de E. coli, tissu intestinal de veau ou crevette arctique), élimine le groupe phosphate présent à l’extrémité 5 ‘d’une molécule d’ADN. (B) Polynucleotide kinase: (provenant de E. coli infectée par le phage T4), qui a l’effet inverse de la phosphatase alcaline, ajoutant des groupes phosphate sur des terminaisons 5 ‘libres.
(C) Terminal désoxynucléotidyl transférase: (à partir de tissu de thymus de veau), ajoute un ou plusieurs désoxyribonucléotides sur l’extrémité 3 ‘d’une molécule d’ADN.
Enzymes pour la coupe de l’ADN : des endonucléases de restriction .
Le clonage de gènes nécessite que les molécules d’ADN soient coupées d’une manière très précise et reproductible. Ceci est illustré par la façon dont le vecteur est coupé pendant la construction d’une molécule d’ADN recombinant. Chaque molécule de vecteur doit être clivée en une seule position, pour ouvrir le cercle de sorte qu’un nouvel ADN puisse être inséré: une molécule qui est coupée plus d’une fois sera cassée en deux ou plusieurs fragments séparés et ne sera d’aucune utilité comme vecteur de clonage. En outre, chaque molécule vectorielle doit être coupée exactement à la même position sur le cercle – comme cela apparaîtra dans les chapitres suivants Le clivage, aléatoire n’est pas satisfaisant. Il doit être clair qu’un type très spécial de nucléase est nécessaire pour effectuer cette manipulation. Souvent, il est également nécessaire de cliver l’ADN qui doit être cloné. Il y a deux raisons à cela.
Premièrement, si l’objectif est de cloner un seul gène, qui peut se composer seulement de 2 ou 3 kb d’ADN, alors ce gène devra être coupé des grosses molécules d’ADN (souvent plus de 80 kb) produites par l’utilisation habile des techniques de préparation décrites au chapitre 3.
Deuxièmement, de grandes molécules d’ADN peuvent avoir à être décomposées simplement pour produire des fragments suffisamment petits pour être portés par le vecteur. La plupart des vecteurs de clonage présentent une préférence pour des fragments d’ADN qui tombent dans une plage de tailles particulière: la plupart des vecteurs à base de plasmide, par exemple, sont très inefficaces au clonage de molécules d’ADN de plus de 8 kb de longueur.
Les endonucléases de restriction purifiées permettent au biologiste moléculaire de découper des molécules d’ADN de manière précise et reproductible pour le clonage de gènes. La découverte de ces enzymes, qui a conduit à des prix Nobel pour W. Arber, H. Smith et D. Nathans en 1978, a été l’une des percées clés dans le développement du génie génétique.
La découverte et la fonction des endonucléases de restriction :
L’observation initiale qui a mené à la découverte d’endonucléases de restriction a été faite au début des années 1950, quand il a été montré que certaines souches de bactéries sont immunisées contre l’infection par bactériophage, un phénomène appelé la restriction contrôlée par l’hôte. Le mécanisme de restriction n’est pas très compliqué, même s’il a fallu plus de 20 ans pour être pleinement compris. La restriction se produit parce que la bactérie produit une enzyme qui dégrade l’ADN du phage avant qu’il ait le temps de se répliquer et de diriger la synthèse de nouvelles particules de phage. Le propre ADN de la bactérie, dont la destruction serait évidemment mortelle, est protégé contre l’attaque car il transporte des groupes méthyle supplémentaires qui bloquent l’action enzymatique dégradante. Ces enzymes dégradantes sont appelées endonucléases de restriction et sont synthétisées par de nombreuses espèces bactériennes, peut-être toutes: plus de 2500 différentes ont été isolées et plus de 300 sont disponibles pour être utilisées au laboratoire.
Trois classes différentes d’endonucléases de restriction sont reconnues, chacune étant distinguée par un mode d’action légèrement différent. Les types I et III sont plutôt complexes et n’ont qu’un rôle limité dans le génie génétique. Par contre les endonucléases de restriction de type II, sont très importantes dans le clonage de gènes.

Les endonucléases de restriction de type II coupent l’ADN à des séquences nucléotidiques spécifiques :
La caractéristique centrale des endonucléases de restriction de type II (que l’on appellera simplement « endonucléases de restriction ») est que chaque enzyme a une séquence de reconnaissance spécifique au niveau de laquelle elle coupe une molécule d’ADN. Une enzyme particulière qui clive l’ADN à la séquence de reconnaissance spécifique n’a aucun effet ailleurs. Par exemple, l’endonucléase de restriction appelée PvuI (isolée de Proteus vulgaris) coupe l’ADN uniquement au niveau de l’hexanucléotide CGATCG. Par contre, une seconde enzyme de la même bactérie, appelée PvuII, coupe au niveau de l’hexanucléotide CAGCTG. De nombreuses endonucléases de restriction reconnaissent les sites cibles hexanucléotidiques, mais d’autres coupent à quatre, cinq, huit ou même des séquences nucléotidiques plus longues. Sau3A (de Staphylococcus aureus souche 3A) reconnaît GATC, et AluI (Arthrobacter luteus) coupe à AGCT. Il existe également des exemples d’endonucléases de restriction avec des séquences de reconnaissance dégénérées, ce qui signifie qu’elles coupent l’ADN à n’importe quel d’une famille de sites apparentés. HinfI (Haemophilus Influenzae souche Rf), par exemple, reconnaît GANTC, donc coupe à GAATC, GATTC, GAGTC et GACTC. Les séquences de reconnaissance pour certaines des endonucléases de restriction les plus fréquemment utilisées sont énumérées dans le tableau ci-dessous.
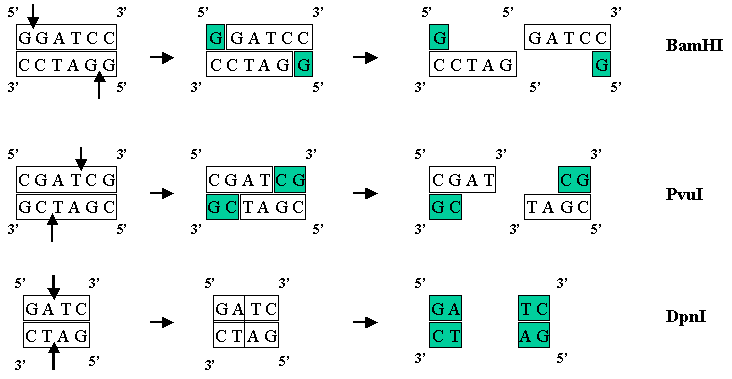

Extrémités coupantes et extrémités collantes:
La nature exacte de la coupe produite par une endonucléase de restriction est d’une importance considérable dans la conception d’une expérience de clonage de gène. De nombreuses endonucléases de restriction font une simple coupure double brin au milieu de la séquence de reconnaissance, ce qui a pour résultat une extrémité franche ou une extrémité affleurante. PvuII et AluI sont des exemples qui donnent une coupure à extrémités franches.

D’autres endonucléases de restriction coupent l’ADN d’une manière légèrement différente. Avec ces enzymes, les deux brins d’ADN ne sont pas coupés exactement dans la même position. Au lieu de cela, le clivage est échelonné, habituellement par deux ou quatre nucléotides, de sorte que les fragments d’ADN résultants présentent des surplombs simples à chaque extrémité. Ceux-ci sont appelés des extrémités collantes ou cohésives, car l’appariement de base entre elles peut coller la molécule d’ADN ensemble encore.

Une caractéristique importante des enzymes terminales collantes est que les endonucléases de restriction avec différentes séquences de reconnaissance peuvent produire les mêmes extrémités collantes. BamHI (séquence de reconnaissance GGATCC) et BglII (AGATCT) sont des exemples – les deux produisent des extrémités collantes GATC. La même extrémité collante est également produite par Sau3A, qui ne reconnaît que le tétranucléotide GATC. Des fragments d’ADN produits par clivage avec l’une ou l’autre de ces enzymes peuvent être joints l’un à l’autre, chaque fragment portant une extrémité collante complémentaire.

La fréquence des séquences de reconnaissance dans une molécule d’ADN :
Le nombre de séquences de reconnaissance pour une endonucléase de restriction particulière dans une molécule d’ADN de longueur connue peut être calculée mathématiquement. Une séquence tétranucléotidique (Par exemple, GATC) doit se produire une fois tous les 44 = 256 nucléotides, et un hexanucléotide (Par exemple, GGATCC) une fois tous les 46 = 4096 nucléotides. Ces calculs supposent que les nucléotides sont ordonnés d’une manière aléatoire et que les quatre nucléotides différents sont présent dans des proportions égales (c’est-à-dire, la teneur en GC = 50%).
En pratique, aucun de ces hypothèses n’est entièrement valable. Par exemple, la molécule d’ADN e, à 49 kb, devrait contenir environ 12 sites pour une endonucléase de restriction avec une séquence de reconnaissance hexanucléotidique . En fait, bon nombre de ces sites de reconnaissance se produisent moins fréquemment (par exemple, six Pour BglII, cinq pour BamHI, et seulement deux pour SalI). En outre, les sites de restriction ne sont pas espacés uniformément le long d’une molécule d’ADN. Si elles l’étaient, alors la digestion avec une endonucléase de restriction particulière donnerait des fragments de tailles approximativement égales. Quand on étudie les fragments produits par BglII, BamHI et SalI, dans chaque cas, il existe des fragments, de taille différente indiquant que les nucléotides (de l’ADN ) ne sont pas ordonnés aléatoirement. La leçon à tirer est que, bien que les mathématiques pourraient donner une idée du nombre de sites de restriction dans une molécule d’ADN, l’ analyse peut fournir la vraie image.